統計学やR言語を勉強していく過程で、R言語のプログラミング的な側面や言語仕様をちゃんと理解したいと思うようになりました。ということでチャンレンジしたのが、『R-Tips
このサイトはいくつかのトピックに分かれているので、自分のRの使い方に合わせて勉強しや少なっています。まだまだ前半戦ですが、特に覚えておきたいコマンドを中心にメモを書いていきます。
(03/22 10:50) 回帰分析部分の説明を追記
Rの便利なメソッド x <- "1245"< span># 中身についての情報を取得 str(x) #=> chr "1245" summary(x) # Length Class Mode # 1 character character # 基本的な情報 y <- c(2, 3, 4, 5)< span>summary(y) # Min. 1st Qu. Median Mean 3rd Qu. Max. # 2.00 2.75 3.50 3.50 4.25 5.00
ベクトルの生成 # aからbまでのベクトルを生成 => a:b 1:10 #=> 1 2 3 4 5 6 7 8 9 10 # (a, b)間をn等分するベクトルを生成 => seq(a, b, length = n) seq(1, 2, length = 10) # 1.000000 1.111111 1.222222 1.333333 1.444444 # 1.555556 1.666667 1.777778 1.888889 2.000000 # aからbまでcずつ増加するベクトルを生成 => seq(a, b, by = c) seq(2, 15, by=3) #=> 2 5 8 11 14 # a:bをc回繰り返すベクトルを生成 => rep(a:b, times = c) rep(1:3, times = 2) #=> 1 2 3 1 2 3 # ベクトル内の値をユニークにする(重複した値を取り除く) => unique(x) unique(c(1,2,4,6,3,3,2,1)) #=> 1 2 4 6 3 # 0をn個並べたベクトルを生成 numeric(5) #=> 0 0 0 0 0 x <- 1:5< span># 要素の並びを逆順にする rev(x) #=> 5 4 3 2 1 y <- 5:1< span># 要素の並び順を整列させる sort(y) #=> 1 2 3 4 5
ベクトル条件 # どれか一つでも条件をみたすものがあるか? any(y > 4) #=> TRUE # すべて条件をみたすか? all(y > 2) #=> FALSE # NULLか否か is.null(NULL) #=> TRUE # NA(データの欠損)か否か is.na(NA) #=> TRUE # NaN(数では表せない)か否か is.nan(0/0) #=> TRUE # 有限か否か is.finite(1) #=> TRUE # 無限か否か is.infinite(1/0) #=> TRUE
ベクトルの中身 x <- 1:5< span># 2-4番目の要素 x[2:4] #=> 2, 3, 4 # 1, 4番目の要素以外を取り出す x[c(-1,-4)] #=> 2, 3, 5 # 3より大きい要素を取り出す x[3 < x] #=> 4, 5 # 1より大きく、4より小さい要素を取り出す x[1 < x & x < 4] #=> 2, 3 y <- x * 2< span># 条件を満たす要素の番号を返す (1:length(y))[1 < y & y < 8] #=> 1, 2, 3
日付 # 日付型に変換 as.Date("2012/08/15") #=> "2012-08-15" as.Date("20130222", format="%Y%m%d") #=> "2013-02-22" # 現在時刻を日付オブジェクトで取得 today <- sys.date()< span>now <- sys.time()< span># 日付の差分 x <- as.date(c("2012 07 01", "2009 08 01"))< span>x[2] - x[1] #=> Time difference of -1065 days
文字列 # 文字列ベクトルの結合 paste(month.abb, 1:8, c("st", "nd", "rd", rep("th",5))) # "Jan 1 st" "Feb 2 nd" "Mar 3 rd" "Apr 4 th" "May 5 th" "Jun 6 th" # "Jul 7 th" "Aug 8 th" "Sep 1 st" "Oct 2 nd" "Nov 3 rd" "Dec 4 th" # 文字列から特定の部分を切り出し substring("defghijklmnopqr", 3, 7) #=> "fghij"
データフレーム # データフレームの作成 sex <- c(rep("f", 3), rep("m", 1))< span>height <- seq(150, 180, length="4)weight <- seq(70, 80, length="4)df <- data.frame(sex, height, weight)< span># sex height weight # 1 F 150 70.00000 # 2 F 160 73.33333 # 3 F 170 76.66667 # 4 M 180 80.00000 # データオブジェクトの行数を返す nrow(df) #=> 4 # summaryでデータフレームの要素の情報を表示 summary(df) # sex height weight # F:3 Min. :150.0 Min. :70.0 # M:1 1st Qu.:157.5 1st Qu.:72.5 # Median :165.0 Median :75.0 # Mean :165.0 Mean :75.0 # 3rd Qu.:172.5 3rd Qu.:77.5 # Max. :180.0 Max. :80.0 # heightで降順に並べ直し sort_list <- order(df$height,decreasing="T)df[sort_list,] # sex height weight # 4 M 180 80.00000 # 3 F 170 76.66667 # 2 F 160 73.33333 # 1 F 150 70.00000 # typeカラムに条件に応じてBIG, MIDDLE, SMALL df$type <- ifelse(height>= 170, "BIG", ifelse(height>=160, "MIDDLE", "SMALL")) # sex height weight type # 1 F 150 70.00000 SMALL # 2 F 160 73.33333 MIDDLE # 3 F 170 76.66667 BIG # 4 M 180 80.00000 BIG # 一律でheight_mmカラムを追加 transform(df, height_mm=df$height*100) # sex height weight type height_mm # 1 F 150 70.00000 SMALL 15000 # 2 F 160 73.33333 MIDDLE 16000 # 3 F 170 76.66667 BIG 17000 # 4 M 180 80.00000 BIG 18000 # 行番号を追加 df$id <- row.names(df)< span># sex height weight type id # 1 F 150 70.00000 SMALL 1 # 2 F 160 73.33333 MIDDLE 2 # 3 F 170 76.66667 BIG 3 # 4 M 180 80.00000 BIG 4 # 行方向の結合 cbind(df, df) # sex height weight type id sex height weight type id # 1 F 150 70.00000 SMALL 1 F 150 70.00000 SMALL 1 # 2 F 160 73.33333 MIDDLE 2 F 160 73.33333 MIDDLE 2 # 3 F 170 76.66667 BIG 3 F 170 76.66667 BIG 3 # 4 M 180 80.00000 BIG 4 M 180 80.00000 BIG 4 # 列方向の結合 rbind(df, df) # sex height weight type id # 1 F 150 70.00000 SMALL 1 # 2 F 160 73.33333 MIDDLE 2 # 3 F 170 76.66667 BIG 3 # 4 M 180 80.00000 BIG 4 # 5 F 150 70.00000 SMALL 1 # 6 F 160 73.33333 MIDDLE 2 # 7 F 170 76.66667 BIG 3 # 8 M 180 80.00000 BIG 4
🐠 サンプルデータセット Rで勉強していく上で、避けて通れないのがサンプルデータの確保です。Rにはデフォルトで複数のデータセットが格納されています。『統計を学びたい人へ贈る、統計解析に使えるデータセットまとめ
🚜 データセットを使った集計・グラフ表示 デフォルトのデータセットのあやめのデータ(iris)を使って、いくつかサンプルを書いていきます。
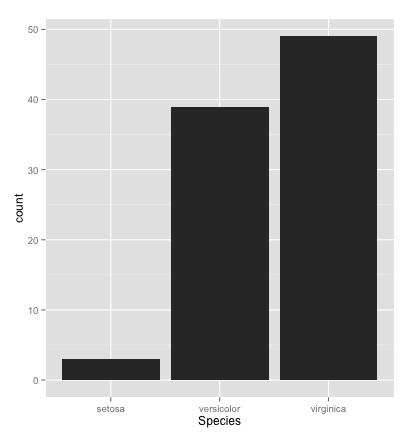
# irisはあやめのデータ・セット width_set <- iris[,c("sepal.length", "petal.length", "species")]< span>width_large <- width_set[width_set$species="=" "versicolor",]< span>mean(width_large$Petal.Length) #=> 4.26 # plyrパッケージの導入 # plyr : データフレームの加工をしやすくするパッケージ install.packages("plyr") library("plyr") # irisのデータからSpeciesをキーにSepal.Length(がくの長さ)の平均を出す data <- ddply(iris, .(species), summarise, sepal.length.average="mean(Sepal.Length))# Species Sepal.Length.Average # 1 setosa 5.006 # 2 versicolor 5.936 # 3 virginica 6.588 # Sepal.Length(がくの長さ)の平均順でならべ直す arrange(data, desc(Sepal.Length.Average)) # Species Sepal.Length.Average # 1 virginica 6.588 # 2 versicolor 5.936 # 3 setosa 5.006 # 棒グラフ # 棒グラフは「月ごとの販売実績」のように書くカテゴリの度数や比率を表示するために利用する index <- iris$sepal.length> 5.5 dat <- iris[index,]< span>xtabs(~Species, data=dat, drop.unused.levels=T) # Species # setosa versicolor virginica # 3 39 49 qplot(Species, data=dat, geom="bar")
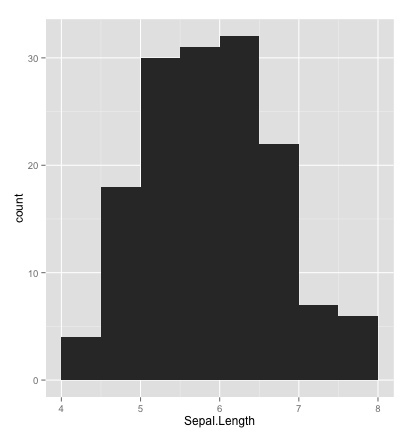
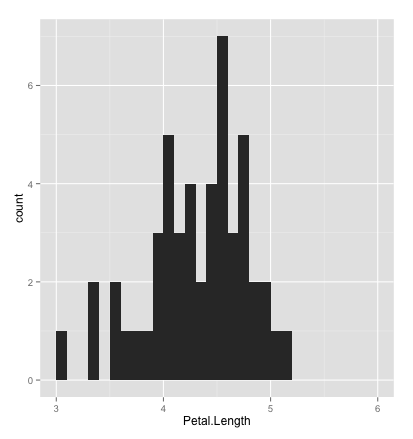
# ヒストグラム # 身長・成績・人口などのように連続量のデータの度数(割合)を表示する qplot(Sepal.Length, data=iris, geom="histogram", binwidth=0.5, xlim=c(4,8))
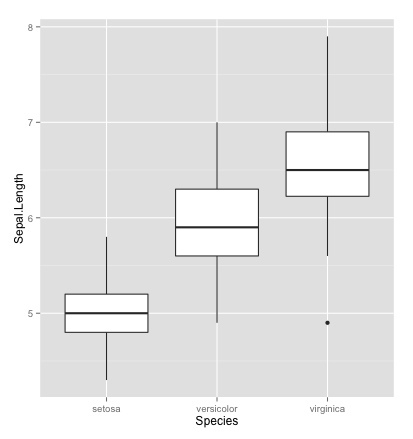
# 箱ひげ図 # カテゴリごとの中央値、箱、ヒゲ、外れ値を表示。ヒストグラムの簡略版 # 1つが複数カテゴリのデータ、もう一つが連続量の場合に用いる qplot(Species, Sepal.Length, data=iris, geom="boxplot")
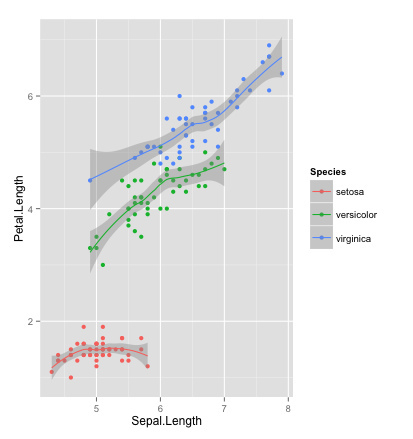
# 散布図 # 2つの変数の値に従って座標平面上に複数の点を表示 # 2つの変数がともに連続量の場合に用いる index <- iris$species !="virginica" < span>dat <- iris[index,]< span>qplot(Sepal.Length, Petal.Length, data=iris, geom=c("point", "smooth"), color=Species)
🐮 サンプルデータを使った単回帰分析 サンプルデータを知らなすぎて、あまり適切な分析ではないですが、手元にある範囲内で。
キーワード 独立変数: 原因となる変数
従属変数: 性質を明らかにしたい側の変数
単回帰分析: 独立変数が1つの分析
重回帰分析: 独立変数が2つ以上の分析
# データ・セットを使った回帰分析のサンプル library(ggplot2) qplot(Petal.Length, data=dat, geom="histogram", bandwith=0.5, xlim=c(3,6))
index <- iris$species="=" 'versicolor'< span>dat <- iris[index,]< span>result <- glm(sepal.length~petal.length, data="dat," family="gaussian)summary(result) # glm(formula = Sepal.Length ~ Petal.Length, family = gaussian, # data = dat) # # Deviance Residuals: # Min 1Q Median 3Q Max # -0.73479 -0.20272 -0.02065 0.26092 0.69956 # # Coefficients: # Estimate Std. Error t value Pr(>|t|) # (Intercept) 2.4075 0.4463 5.395 2.08e-06 *** # Petal.Length 0.8283 0.1041 7.954 2.59e-10 *** # --- # Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1 # # (Dispersion parameter for gaussian family taken to be 0.1173364) # # Null deviance: 13.0552 on 49 degrees of freedom # Residual deviance: 5.6321 on 48 degrees of freedom # AIC: 38.717 # # Number of Fisher Scoring iterations: 2 #=> 回帰式 y = ax + bで a = 2.4075, b = 0.8283 #=> Coefficientsの最後が信頼度。'***'なので高い
🤔 適切な回帰分析式の比較 AIC : 赤池情報量規準 統計モデルの良さを評価するための指標。
統計モデルの比較 こちらも素人のトライですので、ツッコミお待ちしています!
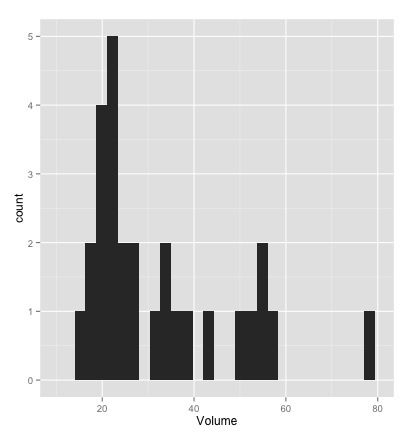
library(ggplot2) # 単純にヒストグラムでプロット qplot(Volume, data=trees, geom="histogram", bandwith=5, xlim=c(10,80))
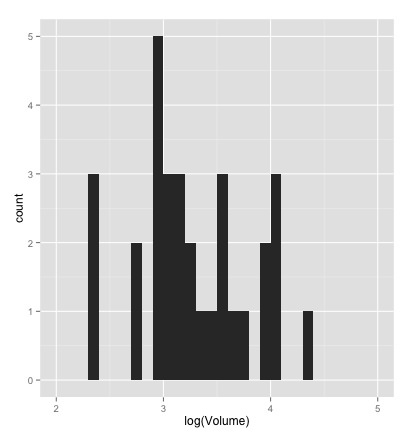
# 対数をとってヒストグラムでプロット qplot(log(Volume), data=trees, geom="histogram", bandwith=0.2, xlim=c(2,5))
本来は正規分布に従いそうなデータですが、サンプルデータを目視する限りは対数正規分布に近そう。
result <- glm(volume~girth, data="trees," family="gaussian)summary(result) # (Intercept) -36.9435 3.3651 -10.98 7.62e-12 *** # Girth 5.0659 0.2474 20.48 < 2e-16 *** # AIC: 181.64 result <- glm(volume~girth, data="trees," family="gaussian(link="log"))summary(result) # (Intercept) 1.340516 0.097224 13.79 2.89e-14 *** # Girth 0.147910 0.005839 25.33 < 2e-16 *** # AIC: 169.14 result <- glm(volume~(girth+height), data="trees," family="gaussian)summary(result) # (Intercept) -57.9877 8.6382 -6.713 2.75e-07 *** # Girth 4.7082 0.2643 17.816 < 2e-16 *** # Height 0.3393 0.1302 2.607 0.0145 * # AIC: 176.91 result <- glm(volume~(girth+height), data="trees," family="gaussian(link="log"))summary(result) # (Intercept) 0.679294 0.258124 2.632 0.01366 * # Girth 0.134163 0.006845 19.601 < 2e-16 *** # Height 0.011144 0.003975 2.804 0.00907 ** # AIC: 163.37 #=> 上記4つのAIC(赤池情報量規準)からみると一番最後の回帰式(対数正規分布で独立変数にGirth, Heightをとる)が最も近い
🐞 ポワソン回帰 ポワソン回帰は離散値かつ、ゼロ以上の範囲のデータを扱うときに利用する。
適切なデータでないんで、雰囲気だけメモ。
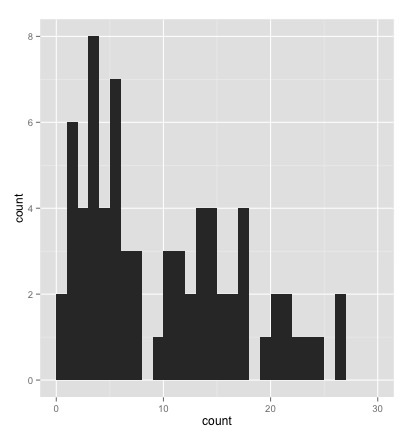
dat <- insectsprays < span>qplot(count, data=dat, geom="histogram", bandwith=1, xlim=c(0,30))
result <- glm(count~spray, data="dat," family="poisson(link="log"))summary(result) # (Intercept) 2.67415 0.07581 35.274 < 2e-16 *** # sprayB 0.05588 0.10574 0.528 0.597 # sprayC -1.94018 0.21389 -9.071 < 2e-16 *** # sprayD -1.08152 0.15065 -7.179 7.03e-13 *** # sprayE -1.42139 0.17192 -8.268 < 2e-16 *** # sprayF 0.13926 0.10367 1.343 0.179 # AIC: 376.59
🗽 独立変数の最適化 複数の独立変数から、Rが効率的なアルゴリズムで自動的にモデル選択をする方法。
適切なサンプルデータで後でリトライする予定…
# 独立変数の最適化 dat <- iris< span>result <- glm(petal.width~sepal.length+sepal.width+petal.length, data="dat," family="gaussian(link="log"))step(result, direction="backward") # Start: AIC=19.47 # Petal.Width ~ Sepal.Length + Sepal.Width + Petal.Length # # Df Deviance AIC # - Sepal.Width 1 9.472 19.337 # 9.355 19.469 # - Sepal.Length 1 12.225 57.615 # - Petal.Length 1 33.293 207.890 # # Step: AIC=19.34 # Petal.Width ~ Sepal.Length + Petal.Length # # Df Deviance AIC # 9.472 19.337 # - Sepal.Length 1 12.371 57.394 # - Petal.Length 1 38.191 226.476 # # Call: glm(formula = Petal.Width ~ Sepal.Length + Petal.Length, family = gaussian(link = "log"), # data = dat) # # Coefficients: # (Intercept) Sepal.Length Petal.Length # -0.3528 -0.2442 0.4752 # # Degrees of Freedom: 149 Total (i.e. Null); 147 Residual # Null Deviance: 86.57 # Residual Deviance: 9.472 AIC: 19.34 # #=> 以上より、Petal.Width = -0.2442*Sepal.Length + 0.4752*Petal.Length + -0.3528 がAICでベスト
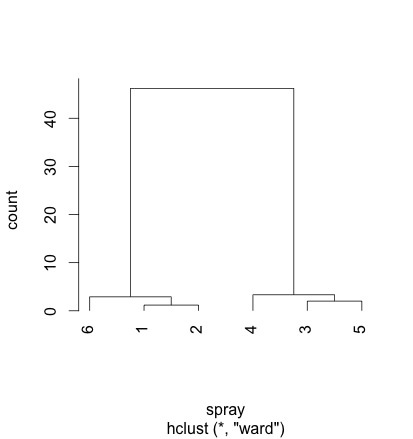
🏈 ユークリッド距離を使ったクラスタリング ユークリッド距離や重心の概念を使ってクラスタリングを行う。
Rに難儀しているので、とりあえず暫定でできたところまで。使い方を覚えたらちゃんと直したい!
library("plyr") dat <- insectsprays< span>dat2 <- ddply(dat, .(spray), summarise, count_ave="mean(count))distw <- dist(dat2, method="euclidean" )< span>clust <- hclust(distw, method="ward" )< span>plclust(clust, hang=-1, xlab="spray", ylab="count")
🍄 参考リンク 「plyrパッケージで君も前処理スタ☆」改め「plyrパッケージ徹底入門」
🎃 あとで読む 統計・機械学習・データマイニングの無料で読めるPDF資料
統計的機械学習 | 中川研究室
一般向けのDeep Learning
統計検定:Japan Statistical Society Certificate
チュートリアル | グローバル消費インテリジェンス寄附講座
🚌 変更来歴 (03/15 10:45) 回帰に関する記述を追記
🖥 VULTRおすすめ
「VULTR VULTR ここ